在本教程中,您将学习有关直方图和密度图的所有信息。
准备好笔记本
和往常一样,我们从设置编码环境开始。
import pandas as pd
import matplotlib.pyplot as plt
%matplotlib inline
import seaborn as sns
print("Setup Complete")选择一个数据集
我们将使用一个包含150种不同花的数据集,或来自三种不同鸢尾属的各50种花的数据集。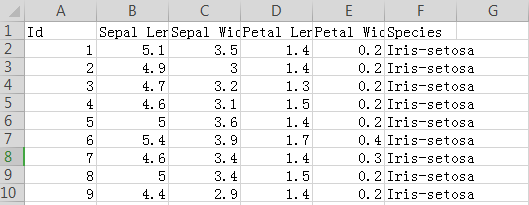
加载并检查数据
数据集中的每一行对应于不同的花。有四种测量方法:萼片的长度和宽度,以及花瓣的长度和宽度。我们也跟踪相应的物种。
# Path of the file to read
iris_filepath = "../input/iris.csv"
# Read the file into a variable iris_data
iris_data = pd.read_csv(iris_filepath, index_col="Id")
# Print the first 5 rows of the data
iris_data.head()输出: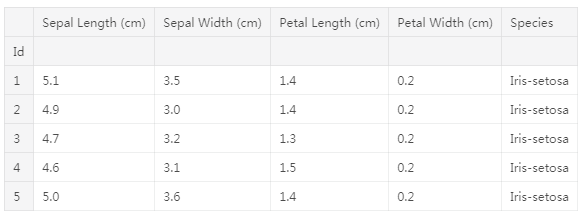
直方图
假设我们想要创建一个直方图来查看鸢尾花的花瓣长度是如何变化的。我们可以用sns.distplot命令来做。
# Histogram
sns.distplot(a=iris_data['Petal Length (cm)'], kde=False)输出:
/opt/conda/lib/python3.6/site-packages/scipy/stats/stats.py:1713: FutureWarning: Using a non-tuple sequence for multidimensional indexing is deprecated; use `arr[tuple(seq)]` instead of `arr[seq]`. In the future this will be interpreted as an array index, `arr[np.array(seq)]`, which will result either in an error or a different result.
return np.add.reduce(sorted[indexer] * weights, axis=axis) / sumval
<matplotlib.axes._subplots.AxesSubplot at 0x7f4abd79f630>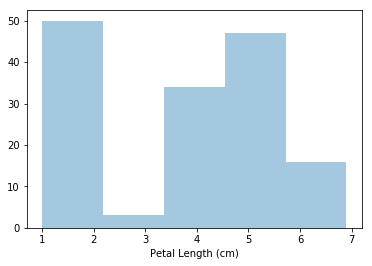
我们使用两个额外的信息自定义命令的行为:
a=选择要绘制的列(在本例中,我们选择了“花瓣长度(cm)”)。- 在创建直方图时,我们总是会提供
kde=False,因为省略它会创建一个稍微不同的图。
密度图
下一种类型的图是内核密度估计(KDE)图。如果您不熟悉KDE图,您可以将它看作一个平滑的直方图。
为了绘制KDE图,我们使用sns.kdeplot命令。将shade=True colors设置为曲线下方的区域(data=具有与上面直方图相同的功能)。
# KDE plot
sns.kdeplot(data=iris_data['Petal Length (cm)'], shade=True)输出:
/opt/conda/lib/python3.6/site-packages/scipy/stats/stats.py:1713: FutureWarning: Using a non-tuple sequence for multidimensional indexing is deprecated; use `arr[tuple(seq)]` instead of `arr[seq]`. In the future this will be interpreted as an array index, `arr[np.array(seq)]`, which will result either in an error or a different result.
return np.add.reduce(sorted[indexer] * weights, axis=axis) / sumval
<matplotlib.axes._subplots.AxesSubplot at 0x7f4acb72c080>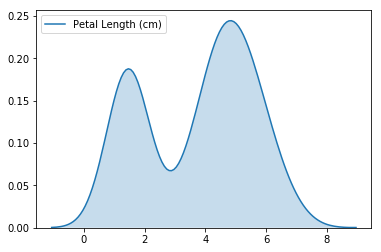
2D KDE图
在创建KDE图时,我们不受限于单个列。我们可以用sns.jointplot命令创建一个二维(2D) KDE图。
在下面的图中,颜色编码显示了我们看到萼片宽度和花瓣长度不同组合的可能性有多大,图中较暗的部分可能性更大。
# 2D KDE plot
sns.jointplot(x=iris_data['Petal Length (cm)'], y=iris_data['Sepal Width (cm)'], kind="kde")输出:
/opt/conda/lib/python3.6/site-packages/scipy/stats/stats.py:1713: FutureWarning: Using a non-tuple sequence for multidimensional indexing is deprecated; use `arr[tuple(seq)]` instead of `arr[seq]`. In the future this will be interpreted as an array index, `arr[np.array(seq)]`, which will result either in an error or a different result.
return np.add.reduce(sorted[indexer] * weights, axis=axis) / sumval
<seaborn.axisgrid.JointGrid at 0x7f4ab9e2f4a8>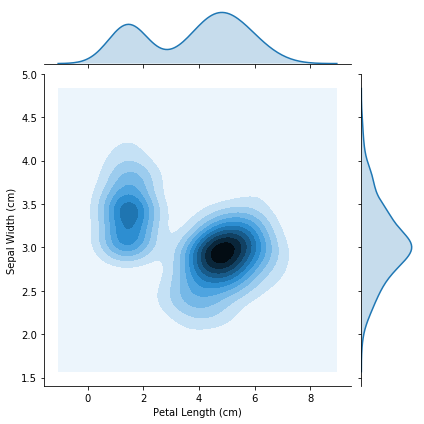
请注意,除了中心的2D KDE图外,
- 图顶部的曲线是x轴上数据的KDE图(在这种情况下,iris_data [‘花瓣长度(cm)’]),以及
- 图右侧的曲线是y轴上数据的KDE图(在本例中为iris_data [‘Sepal Width(cm)’])。
彩色图
在本教程的下一部分中,我们将创建一些图来了解物种之间的差异。为此,我们首先将数据集分成三个单独的文件,每个物种一个。
# Paths of the files to read
iris_set_filepath = "../input/iris_setosa.csv"
iris_ver_filepath = "../input/iris_versicolor.csv"
iris_vir_filepath = "../input/iris_virginica.csv"
# Read the files into variables
iris_set_data = pd.read_csv(iris_set_filepath, index_col="Id")
iris_ver_data = pd.read_csv(iris_ver_filepath, index_col="Id")
iris_vir_data = pd.read_csv(iris_vir_filepath, index_col="Id")
# Print the first 5 rows of the Iris versicolor data
iris_ver_data.head()输出: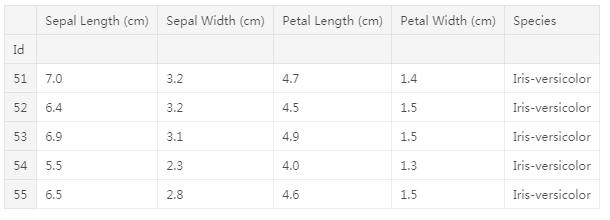
在下面的代码单元格中,我们使用sns.distplot命令为每个物种创建一个不同的直方图(如上所示)三次。我们使用label=设置每个柱状图将如何显示在图例中。
# Histograms for each species
sns.distplot(a=iris_set_data['Petal Length (cm)'], label="Iris-setosa", kde=False)
sns.distplot(a=iris_ver_data['Petal Length (cm)'], label="Iris-versicolor", kde=False)
sns.distplot(a=iris_vir_data['Petal Length (cm)'], label="Iris-virginica", kde=False)
# Add title
plt.title("Histogram of Petal Lengths, by Species")
# Force legend to appear
plt.legend()输出:
/opt/conda/lib/python3.6/site-packages/scipy/stats/stats.py:1713: FutureWarning: Using a non-tuple sequence for multidimensional indexing is deprecated; use `arr[tuple(seq)]` instead of `arr[seq]`. In the future this will be interpreted as an array index, `arr[np.array(seq)]`, which will result either in an error or a different result.
return np.add.reduce(sorted[indexer] * weights, axis=axis) / sumval
<matplotlib.legend.Legend at 0x7f4ab9cab550>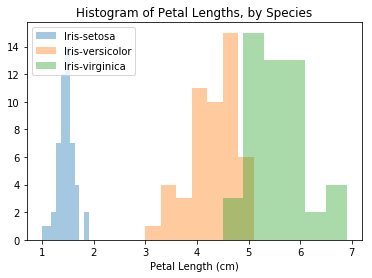
在这种情况下,图例不会自动出现在图标中。要强制它显示(对于任何图标类型),我们可以使用pl.legend()。
我们还可以使用sns.kdeplot为每个物种创建一个KDE图(如上所述)。同样,label=用于设置图例中的值。
# KDE plots for each species
sns.kdeplot(data=iris_set_data['Petal Length (cm)'], label="Iris-setosa", shade=True)
sns.kdeplot(data=iris_ver_data['Petal Length (cm)'], label="Iris-versicolor", shade=True)
sns.kdeplot(data=iris_vir_data['Petal Length (cm)'], label="Iris-virginica", shade=True)
# Add title
plt.title("Distribution of Petal Lengths, by Species")输出:
/opt/conda/lib/python3.6/site-packages/scipy/stats/stats.py:1713: FutureWarning: Using a non-tuple sequence for multidimensional indexing is deprecated; use `arr[tuple(seq)]` instead of `arr[seq]`. In the future this will be interpreted as an array index, `arr[np.array(seq)]`, which will result either in an error or a different result.
return np.add.reduce(sorted[indexer] * weights, axis=axis) / sumval
Text(0.5, 1.0, 'Distribution of Petal Lengths, by Species')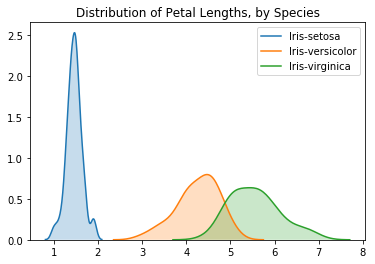
可以在图中看到的一个有趣的模式是植物似乎属于两组中的一组,其中Iris versicolor和Iris virginica似乎具有相似的花瓣长度值,而Iris setosa本身属于一个类别。
事实上,根据这个数据集,我们甚至可以通过观察花瓣长度将任何虹膜植物分类为虹膜(与虹膜鸢尾(Iris versicolor)或虹膜鸢尾(Iris virginica)相对):如果鸢尾花的花瓣长度小于 2厘米,它最有可能是Iris setosa!
本地运行代码:
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
# Path of the file to read
iris_filepath = "data-for-datavis/iris.csv"
# Read the file into a variable iris_data
iris_data = pd.read_csv(iris_filepath, index_col="Id")
# Print the first 5 rows of the data
print(iris_data.head())
# Histogram
sns.distplot(a=iris_data['Petal Length (cm)'], kde=False)
# KDE plot
sns.kdeplot(data=iris_data['Petal Length (cm)'], shade=True)
# 2D KDE plot
sns.jointplot(x=iris_data['Petal Length (cm)'], y=iris_data['Sepal Width (cm)'], kind="kde")
plt.show()
# Paths of the files to read
iris_set_filepath = "data-for-datavis/iris_setosa.csv"
iris_ver_filepath = "data-for-datavis/iris_versicolor.csv"
iris_vir_filepath = "data-for-datavis/iris_virginica.csv"
# Read the files into variables
iris_set_data = pd.read_csv(iris_set_filepath, index_col="Id")
iris_ver_data = pd.read_csv(iris_ver_filepath, index_col="Id")
iris_vir_data = pd.read_csv(iris_vir_filepath, index_col="Id")
# Print the first 5 rows of the Iris versicolor data
print(iris_ver_data.head())
# Histograms for each species
sns.distplot(a=iris_set_data['Petal Length (cm)'], label="Iris-setosa", kde=False)
sns.distplot(a=iris_ver_data['Petal Length (cm)'], label="Iris-versicolor", kde=False)
sns.distplot(a=iris_vir_data['Petal Length (cm)'], label="Iris-virginica", kde=False)
# Add title
plt.title("Histogram of Petal Lengths, by Species")
# Force legend to appear
plt.legend()
plt.show()
# KDE plots for each species
sns.kdeplot(data=iris_set_data['Petal Length (cm)'], label="Iris-setosa", shade=True)
sns.kdeplot(data=iris_ver_data['Petal Length (cm)'], label="Iris-versicolor", shade=True)
sns.kdeplot(data=iris_vir_data['Petal Length (cm)'], label="Iris-virginica", shade=True)
# Add title
plt.title("Distribution of Petal Lengths, by Species")
plt.show()
在线练习地址:https://www.kaggle.com/xugj520/exercise-distributions/edit
